2.59 MB
242.90MB
11.0 MB
82.1MB
59M
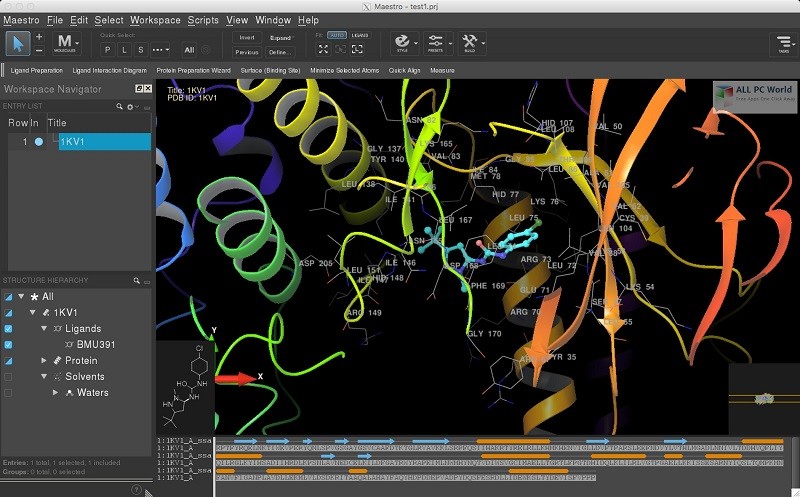
薛定谔软件是学习医学结构的辅助工具。用户可以通过各种技术快速绘制模型,并提供不同的仿真环境供用户观察研究。
薛定谔2免费版是一个专业的药物研究设备软件。该软件的目的是为用户提供强大的功能和最先进的技术,设计和开发药物所需的一切,以及分子建模、药物设计和制造的研究人员。可以快速执行。就是为用户提供舒适的服务。相关环境材料化学结构的快速构建提供了一个简单友好的环境,支持各种相关的计算操作。一般来说,分子和化学结构建模,生物和生物结构建模,图形渲染和数值变化建模,图形和简单的应用环境,不同材料在不同环境条件下的行为。包括评估和分析,原子研究。对水位的物质和模型结构进行各种化学计算,数据显示在各种表格和图形中。
1 .生物制剂试剂盒
从头开始设计,这种易于使用的新试剂盒是第一套完整的所有工具,这对生物制剂、抗体和蛋白质的建模非常重要。
2 .材料套件
这种创新的新套件为基于量子力学的化学系统模拟提供了强大的多功能工具。它可以应用于特殊的化学和材料科学系统。
3 .发现信息学套件
协作药物设计的下一代平台允许医学化学家、生物学家、建模师、IT专业人员和其他人的跨学科团队实时共享、查看和管理数据。
4,皮摩尔
PyMOL是由用户发起的基于开源的分子可视化系统。通过购买维护和支持订阅来支持此类开放、有效且经济实惠的软件的开发。
1.解压缩机箱后,单击setup.exe开始安装,并选择要安装的位置。
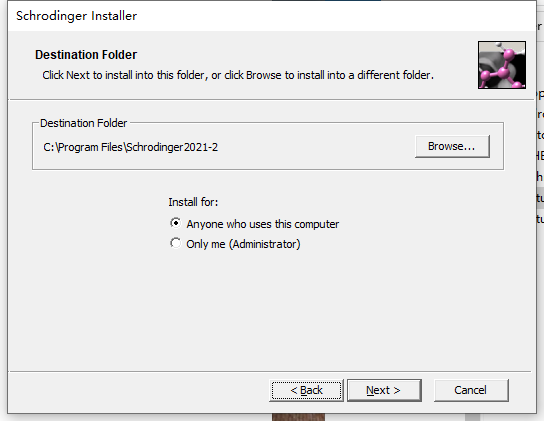
2.检查完所有内容后,单击下一步。
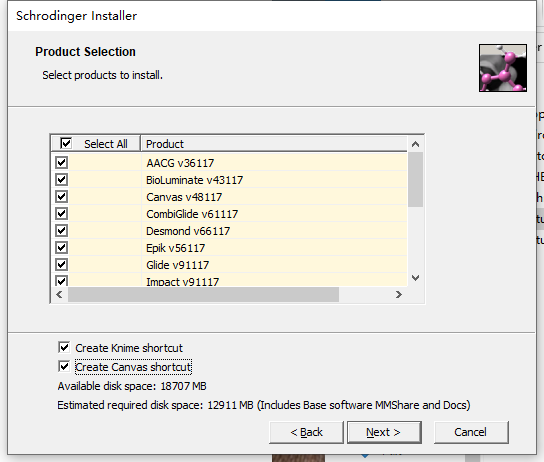
3.单击开始安装,等待安装完成。
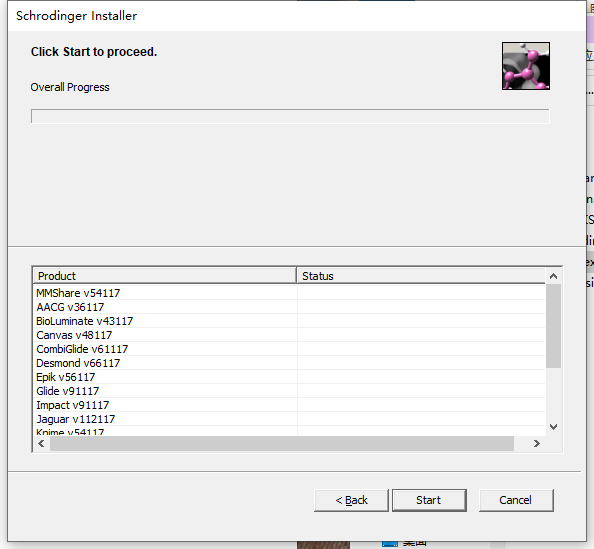
4.单击“确定”完成安装。
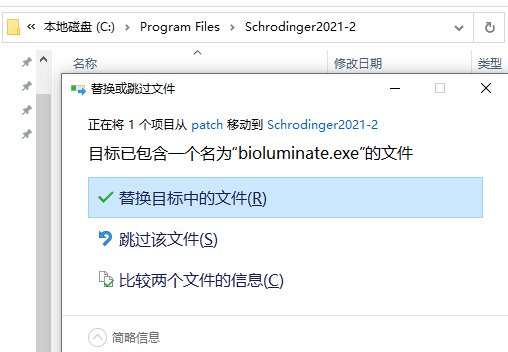
5.将破解文件薛定谔2020-3的文件夹复制到安装目录。默认路径是C:program files Schrodinger 2020-3。点按以替换目的位置中的文件,以便您可以自由使用它。
1 .快速轻松地执行手动或引导线优化
通过自动预测蛋白质配体复杂形状的“设计方法”
使用自定义R组库和属性过滤器来调整项目的化学属性。
使用基于属性的雷达图(MW、logP、HBA、HBD、PSA)快速评估分子适用性。从PT添加用户属性。
提交自己喜欢的设计,根据与FEP+的相似度进行排名,后期处理后发布到Live Design,或者提交到Excel。
2,多序列查看器
支持序列比对和比对,比对方法设置和参数调整选项。
生成成对比较矩阵。根据相似性、同一性或保留性比较整个序列或选定的色谱柱。
将不同来源的信息分层,以增强对序列间差异或相似性的解释。
您可以计算丰富的内建序列描述符,并在序列旁边的指示条中显示值。
这些属性允许您灵活地上传用户生成的描述符和颜色序列。
同源建模:一个引导性的逐步结构预测工作流,带有一个复选标记来指示已完成的步骤。
轻松链接和取消链接序列和结构。
基于全序列比对或结合位点比对的比对结构
一键开关分裂链表示和连接链表示
将连续的颜色应用到您的工作区结构,反之亦然。
3,OPLS3e力场
使用默认的Schrodinger-ANI选项可以加快通过FFBGUI获得的拟合速度。
4、FEP+
集成力场发生器+FEP工作流程
用于快速查看环形边缘纹理/热区数据。
能够清除所有/选定的实验数据
UX显示核心SMARTS的改进
消除多余的SMARTS模式
突出显示与核心SMARTS匹配的节点。
5、分子动力学
移除Desmond CPU
6,混合溶剂MD(MxMD)
包括匹配每个热点的探针结构。
7,共价配体对接
在优化步骤中应用用户定义的约束。
支持原子名称中包含的结合原子(核酸)
8,配体对接
将机器学习与glide对接测试版结合,应用主动学习glide对接一个拥有数十亿化合物的庞大库。
9 .宏
(可选)通过输入文件向宏旋转脚本提供输入。
可以连接和切割分子的融合蛋白肽接头、ADC接头和融合二聚体配体的设计(仅命令行)
10 .停靠
在不影响精度的情况下,IFD-MD的速度提高了约5倍。
XI药理学模型
简化create_hypoConsensus脚本的应用,允许脚本读取*。Phypo格式。
12,配体排列
参考配体的规格可以通过寻找参考配体来改进。
修复了几个错误(如回溯,工作启动失败)
减少了基于flex_align的作业生成的默认追随者的默认数量,以加快计算速度。
添加工具提示
十三。枚举
通过包含描述和名称来改进名称响应搜索。
14,化学信息学
使用画布指纹或属性创建和应用Kohonen自组织地图-Beta
为应用程序创建单独的分类和连续的QSAR模型,并完全控制应用程序和机器学习测试版的功能。
使用kPLS、MLR、PCR、贝叶斯或递归分区机器学习技术创建和创建预测。
使用各种画布指纹、原子和分子描述符或用户提供的描述符创建机器学习模型。
可视化kPLS模型预测原子贡献
或者自动创建测试/训练集分割。
15 .基于经验和质量控制的PKa预测
允许捷豹pKa常规和定制OPLS3e力场执行构象搜索
16、QM / MM (QSite)
站点Q现在支持OPLS3e力场。
17、量子力学
如果要用Spectrum_align.py对齐VCD光谱,可以选择使用可靠的峰值。
计算特定压力范围内的热化学性质
链式互变异构可以在AutoConf.py工作流中生成和评分。
。宇宙遥感计算的宇宙文件
从命令行进行批处理计算现在,您可以选择输出并分组失败的子作业,以进一步简化该过程。
18 .结合位点的特征
新的kinase _ conservation _ analysis . py脚本用于激酶结合位点残基的保守分析。
返回一个独特的残基对,优化小分子的选择性。
原子级功能注释整个基因家族中最独特的残基对。
19、蛋白质同源建模
命令行支持更好地控制所有主要优化作业(循环预测、侧链预测、结合位点优化等)的最小化参数。).)
20、低温电磁学
使用PHENIX/OPLS3e自动扫描约束权重以确定理想权重。
x/opls3E-提高稳定性,支持多种互补格式,支持编写PDB文件。